| Voltar Teoria | Seguinte: Estrutura Secundária
|
Página Principal Biomédica, MEFT |
|
|
Estrutura |
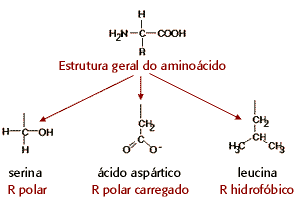
Os aminoácidos apresentam uma estrutura geral que consiste num grupo amino, um grupo carboxílico e uma cadeia lateral R, de dimensão e características variáveis, ligados a um carbono saturado (Cα). Podem ser encontrados 20 diferentes aminoácidos em proteínas.

Propriedades |
A estrutura tridimensional de uma proteína é determinada pela sequência dos aminoácidos constituintes, mais concretamente por interacções não covalentes como ligações de hidrogénio, interacções electrostáticas e hidrofóbicas, empilhamento pi ou, no caso da prolina, por efeitos geométricos impostos pelo aminoácido. É importante assim analisar como as caracteristicas dos aminoácidos determinam estas interacções.
Dimensão
Se imaginarmos uma proteína como um puzzle tridimensional, a
importância da dimensão dos aminoácidos é
imediata. O interior de uma proteína tem uma densidade
comparavel
à de sólidos orgânicos, ou seja, o empacotamento da
proteína é um empacotamento compacto. Assim uma
mutação que substitua uma cadeia lateral pequena por uma
de maiores dimensões pode conduzir à
destabilização da proteína.
Carga
Os aminoácidos Asp, Glu (mononegativos), Lys e Arg
(monopositivos) apresentam-se ionizados na maioria das
condições fisiológicas; dependendo do ambiente
local a histidina pode apresentar-se com uma carga positiva ou na
forma neutra.
A interacção entre duas cadeias laterais com cargas
opostas designa-se por ponte salina ( salt bridge ) existindo numa
proteína típica uma ponte salina por aproximadamente
30 resíduos.
Polaridade
Para além de estabelecerem interacções de van der
Waals, as cadeias laterais ionizadas ou polares participam em
ligações de hidrogénio entre si, com a cadeia
principal ou com o solvente, através dos respectivos grupos
funcionais:
grupo hidroxilo de Ser,Thr e Tyr;
grupo carboxílico de Asp e Glu;
grupo amida de Asn e Gln;
os dois azotos do grupo imidazole da histidina, consoante estejam ou
não protonados, podem funcionar como aceitadores ou doadores de
hidrogénios;
grupo guanadinio da arginina;
grupo amina da lisina;
finalmente o triptofano pode participar numa ligação de
hidrogénio estabelecida pelo átomo de azoto.
Hidrofobicidade
As cadeias laterais alifáticas dos aminoácidos Ala, Val,
Leu and Ile (and Gly) apresentam características
hidrofóbicas. Uma característica geral de
proteínas
globulares consiste na localização de resíduos
hidrofóbicos no interior da proteína enquanto os
resíduos polares ou carregados se situam à
superfície da proteína. A hidrofobicidade é um
factor muito importante na estabilidade de uma proteína, sendo
atribuído um papel relevante ao efeito hidrofóbico no
folding de proteínas.
Os aminoácidos contendo enxofre podem apresentar igualmente
características hidrofóbicas, em especial no caso da
cisteína envolvida na formação de pontes de
enxofre. A cadeia lateral da fenilalanina é fortemente
hidrofóbica. De igual forma o triptofano e a tirosina apresentam
um carácter parcialmente hidrofóbico, não obstante
a presença do átomo de azoto ou do grupo -OH.
Aromaticidade
Os aminoácidos com cadeias laterais aromáticas,
fenilalanina, tirosina, histidina e triptofano participam num tipo de
interacção designado por empilhamento pi (pi stacking).
Cadeias laterias com conformações especiais
Impedimento estereoquímico ( ou ausência dele) implicam
que Pro e Gly são muito importantes na
determinação da conformação da cadeia
peptídica.
Na tabela seguinte são apresentadas as propriedades
dos aminoácidos de acordo com as suas
caracteristicas: com carga positiva , com carga negativa , polares, alifáticos (apolares), e aromáticos. Os aminoácidos
essenciais estão assinalados com um asterisco (*). O
volume
apresentado na tabela é calculado de acordo com A.A.
Zamyatin, Protein Volume in Solution, Prog. Biophys. Mol. Biol.
24(1972)107-123.
Uma descrição exaustiva das propriedades de
aminoácidos pode ser encontrada em Amino Acid Repository,
mantido por Image
Library of Biological Macromolecules, Universidade de Jena.
| Aminoácido | Símbolo | Estrutura |
Cadeia Lateral | Volume |
|
|
|
| Alanina | A, Ala |
|
-CH3 | 88.6 | 6.00 | - | 89.09 |
| Arginina | R, Arg |
|
173.4 | 11.15 | 12.0 | 174.20 | |
| Asparagina | N, Asn |
|
-CH2-CO NH2 | 114.1 | 5.41 | - | 132.12 |
| Ác. Aspártico | D, Asp |
|
-CH2-COO - | 111.1 | 2.77 | 4.4 | 133.10 |
| Cisteína | C, Cys |
|
-CH2-SH | 108.5 | 5.02 | 8.5 | 121.15 |
| Glutamina | Q, Gln |
|
-(CH2)2-C ON2 | 143.8 | 5.65 | - | 146.15 |
| Ác. Glutâmico | E, Glu |
|
-(CH2)2-C OO- | 138.4 | 3.22 | 4.4 | 147.13 |
| Glicina | G, Gly |
|
-H | 60.1 | 5.97 | - | 75.07 |
| Histidina* | H, His |
|
153.2 | 7.47 | 6.5 | 155.16 | |
| Isoleucina* | I, Ile |
|
166.7 | 5.94 | - | 131.17 | |
| Leucina* | L, Leu |
|
166.7 | 5.98 | - | 131.17 | |
| Lisina* | K, Lys |
|
-(CH2)4- NH3+ | 168.6 | 9.59 | 10.0 | 146.19 |
| Metionina* | M, Met |
|
-(CH2)2-S-CH3 | 162.9 | 5.74 | - | 149.21 |
| Fenilalanina* | F, Phe |
|
189.9 | 5.48 | - | 165.19 | |
| Prolina | P, Pro |
|
112.7 | 6.30 | - | 115.13 | |
| Serina | S, Ser |
|
-CH2-OH | 89.0 | 5.68 | - | 105.09 |
| Treonina* | T, Thr |
|
116.1 | 5.64 | - | 119.12 | |
| Triptofano* | W, Trp |
|
227.8 | 5.89 | - | 204.23 | |
| Tirosina | Y, Tyr |
|
193.6 | 5.66 | 10.0 | 181.19 | |
| Valina* | V, Val |
|
|
140.0 | 5.96 | - | 117.15 |
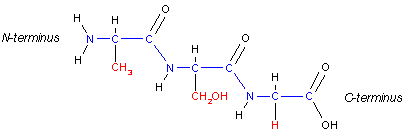
Ligação peptídica |
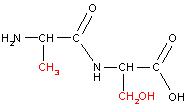
A formação de peptídeos dá-se por
reacção de polimerização de
condensação de aminoácidos. A
ligação
peptídica forma-se entre o átomo de carbono (C) do
grupo carboxílico e o átomo de azoto (N) do grupo amina
com eliminação de água. As duas extremidades da
cadeia assim formada são designadas por "terminal amino
" e " terminal carboxílico" ou N- terminus e
C-
terminus.

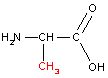
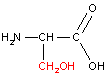
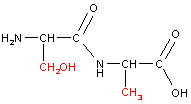
| Aminoácidos |
Peptídeos |
||
| Alanina |
Serina |
Ala-Ser (AS) |
Ser-Ala (SA) |
 |
 |
 |
 |
Ângulos de diedro |
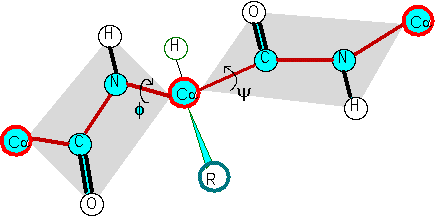
A ligação peptídica apresenta um carácter duplo parcial que inibe a rotação em torno da ligação peptidica e os quatro átomos O,C,N e H definem um plano, o plano peptídico. Uma cadeia polipeptídica pode ser considerada como um conjunto de planos que podem rodar en torno das ligações N-Cα ou C(carbonilo)-Cα .
Na figura seguinte é ilustrado o plano peptidico e os ângulos de diedro (ou torsão) φ e ψ. A vermelho é representado o esqueleto da cadeia peptídica. Estes ângulos estão restritos a determinados valores. A representação gráfica da distribuição destes ângulos numa proteína é designada por Ramachandran plot e é uma forma de aferir a qualidade de uma estrutura.
A conformação do esqueleto de uma cadeia peptidica é completamente descrita por três ângulos de torsão: φ, ψ e Ω, em que o ângulo Ω toma habitualmente os valores 0º e 180º para as configurações cis e trans, respectivamente.
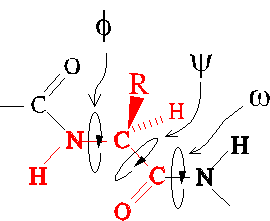
A conformação trans de uma ligação peptidica é mais favorável e mais comum em relação ao confórmero cis devido a impedimento estereoquímico entre cadeias laterais de resíduos adjacentes como é ilustrado na figura seguinte:
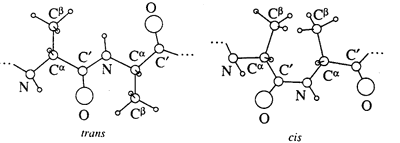
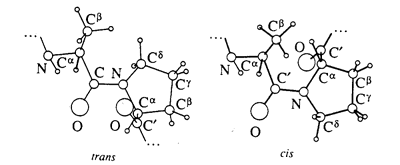
No entanto, para a prolina as configurações cis e trans apresentam energias equivalentes, pelo que é mais habitual encontrar uma prolina cis numa estrutura de uma proteína que qualquer outro aminoácido.
| Página Principal Biomédica, MEFT | Seguinte: Estrutura Secundária | Voltar Teoria |